Genetic association studies(遺伝子関連研究)とは、遺伝子を暴露因子に、とある疾患をアウトカムにして、よく疫学研究で使用されるデザイン(例えば、case-control study)を利用して解析する研究のことをいいます。
Linkage analysisとは種類の異なる研究デザインという点に注意が必要です。
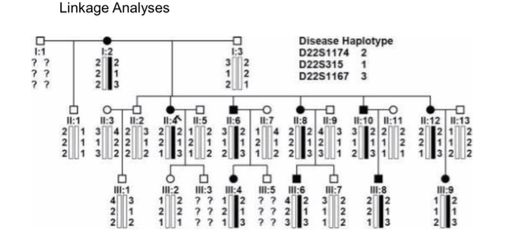
前回まではlinkage analysisについて解説してきました。

Linkage analysisの特徴は、
- 家系図+遺伝子の情報を使い
- 疾患の原因となる遺伝子や染色体の場所を特定する
といった特徴がありました。
この研究の欠点もいくつかありますが、例えば、
- 統計学的な検定力が弱い傾向にある
- 家族や親族のデータが揃わないことが多い
などがあげられます。
このため、疫学研究でよく使用されるケース・コントロール研究などを利用したデザインがあり、これをgenetic association studyと呼んでいます。
Genetic association studyの特徴
Linkage analysisと比較して、genetic association studyの特徴は
- 統計学的な検定力が強い傾向にある
- Family studyが実現不可能の場合でも行える
といった利点があります。
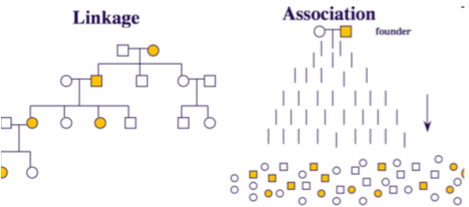
以下の図がLinkage studyとgenetic association studyの違いとして分かりやすいと思います。
Genetic association studyの研究デザインについて
研究デザインですが、大きく分けて、
- Family based study: case-trio studyやその応答
- Case-control study
の2つがあります。
Case-Parent Trio studyについて
Case-parent trio studyは前回説明しているので、簡単に説明します。
図としては、以下のように考えると分かりやすいです。
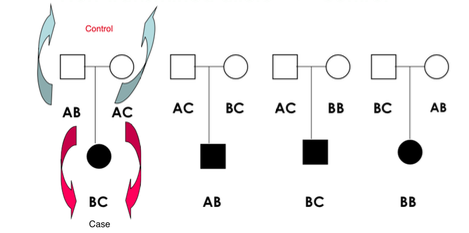
- 両親から受け継いだ遺伝子 (一番左:AA)
- 両親から受け継がなかった遺伝子(一番左:BC)
の両者を比較する研究です。

このペアがいくつあるかを計算します。
特に大事なのはdiscordant pairであるbとcになります。これはmatched case-control studyなどでも同じ考え方ですよね。
Case-parent trio studyの利点と欠点
この研究にも利点と欠点があります。まず利点としては、
- その家庭でのデータを使うのでシンプル
- Population stratificationに影響されない
- Sensitivityが高い
などが挙げられます、
一方で、欠点ですが、
- Population association (Linkage disequilibrium)はない限り、疾患とmarkerの関連性を検出することができない
- 必ずしも家族全員が参加してくれるとは限らない
などがあげられます。
Case-control studyの場合

Case-control studyでgenetic association studyを行う場合、caseとcontrolはアウトカムの発症があるor ないで決まります。
一方で、exposureは遺伝子になります。
どのような遺伝子にするかですが、大きく分けて2通りあり、
- Candidate gene approach
- Genome-wide approach
となります。
後者は少し長くなるので、今回は簡単に触れて終わろうと思います。
Candidate gene approachについて
Candidateは「候補」という意味ですが、既に疾患を起こすメカニズムから考えて候補となる遺伝子が分かっている状態で行う研究です。
Genome-wide approachについて
Genome-wide approachについては、candidate gene approachとは対照的で、詳細なメカニズムなどや候補となる遺伝子が分かっていない状況で、網羅的に検証する方法です。
このため、一度に大量のデータを集めて遺伝的なばらつきをみていきます。
使用するgenetic markerについて
Genetic markerについてですが、対象とする疾患、候補となる遺伝子が判明しているか否かによって異なります。例として、
- SNPs (single nucleotide poltmorphisms)
- Microsatellite
- Copy number variants
などがあります。
*Genome-wide association study (GWAS)については、次回に詳しく説明します。
Linkage analysisとの違い
Genetic association studyですが、linkage analysisとは異なり、特定のalleleと疾患との関連性を検証するのが目的です。
つまり、linkage analysisのように、疾患の原因遺伝子の場所を完全に特定することは目的としていない点に留意しておく必要があります。
CDCVとCDRVについて
CDCVやCDRVと聞いてもあまりピンとこない方が多いと思いますが、それぞれは
- Common disease/common variant (CDCV) hypothesis
- Common disease/rare variant (CDRV) hypothesis
のことを言います。この2つの仮説は遺伝疫学をする上で知っておいたほうが良いでしょう。
Common disease/common variant (CDCV) hypothesis
CDCV hypothesisは、『ありふれた疾患で、それが遺伝と関係している場合は、その原因突然変異は家系が異なっていても(つまり、どのような母集団でも)共通のものが見つかることが多いであろう』という予測から成る仮説です。
この仮説は、candidate gene approachでも、genome wide association studyでも、前提とされる考え方でした。
Common disease/rare variant (CDRV) hypothesis
近年、CDRV hypothesisも提唱されています。
この仮説では、『ありふれた疾患が遺伝と関係している場合、稀なDNA sequenceのばらつきが見つかることが多いであろう』という予測からなる仮説です。
こちらの仮説は、DNA sequence(exome sequenceも含む)のデータなどで使用される仮説で、近年は新しい仮説や統計手法が開発されています。
Genetic associationの解釈
Linkage analysisと異なり、genetic associationの解釈はやや複雑です。
一般論ですが、仮に統計学的に”genetic association”を認めた場合、
- Direct causal relationship
- Indirect association due to LD (linkage disequilibrium)
- False positive due to multiple comparisons
- MarkerのHardy-Weignberg equilibriumからの偏り
- Population stratification
などが原因として考えられます。
Direct causal relationshipについて
ここでいう“Direct”の意味ですが、genetic associationが認められたallele自身が、疾患の発症(phenotype)と直接の因果関係があることを言います。
非常に強い言葉ですが、よほどの根拠が揃っていなければ”direct causal relationship”というのは難しいです。
(*注:Causal mediation analysisのdirect vs. indirectとは意味が異なります)
Indirect association due to LD
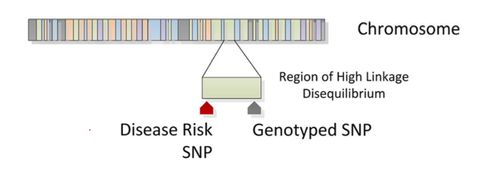
SNPsやMicrosatelliteで判明したalleleと疾患との関連性のことを言います。ここで検出されたAlleleは原因遺伝子の近くにあるだけで、疾患の原因とは言えない場合を”indirect”と言います。
LD (linkage disequilibrium) について
ここで少し脱線して、LD (linkage disequilibrium) について解説していきます。
LDとは、alleles同士のnon-random association(ランダムでない関連性)のことを言います。
LDの計測方法について
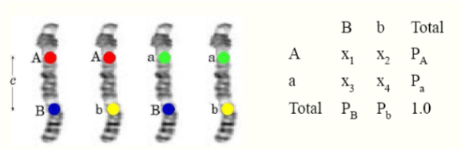
LDの計測ですが、
- D = x1– PA x PB
となります。(t)を世代とすると、Dは以下のようになります:
- D(t)= (1 – c)t D(0)
ここでいうcは、recombination distanceです。例えば、c = 1%, t = 69とすると、
- (1 – c)t = (0.99)69 = 0.50
となり、69世代で50%のLDが減少することを意味します。
つまり、世代を経れば経るほど、LDは減少していきます。
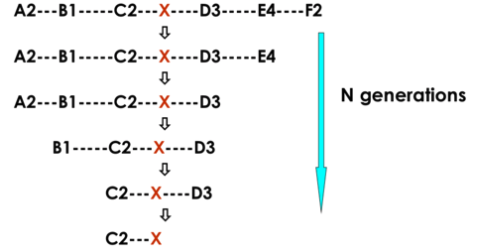
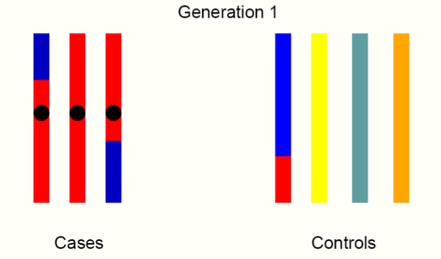
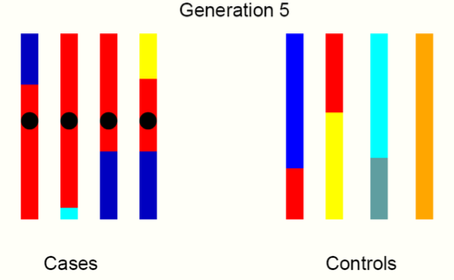
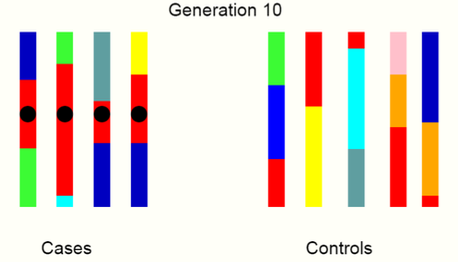
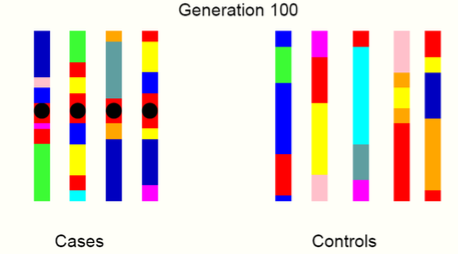
その結果、とある疾患を世代を追っていくことで、特定の原因遺伝子にたどり着くヒントになります。イメージとしては、以下のようになります。
最初
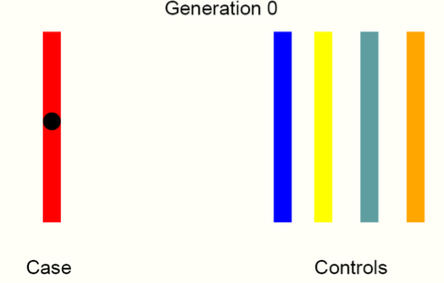
第1世代
第5世代
第10世代
第100世代
LD plotについて
いきなり論文でLD plotを見ても分かりづらいと思うので、まずは例を挙げて解説していきましょう。
LD plotでは、Linkage disequilibriumという距離的な指標を使って、2点の遺伝的な関連性を示しています。
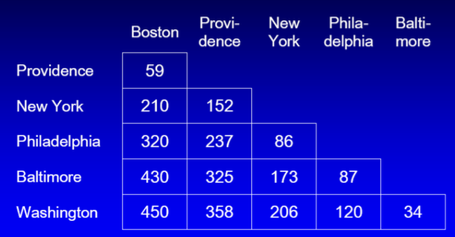
まずは、地理的な例で考えてみましょう。
例えば、アメリカの東海岸には、
- Providence
- NY
- Philadelphia
- Baltimore
- Washington
などがあります。それぞれの距離をmileで記載すると以下のようになります。
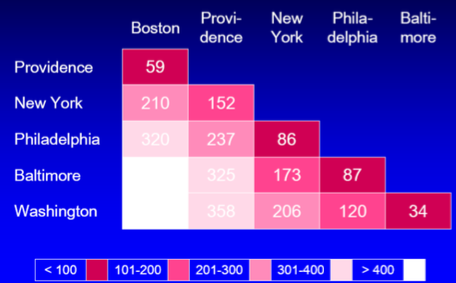
数字だけでみていると辛いので、これを色付けしてみましょう。近いところを濃い赤に、遠いところを白にしてみましょう。
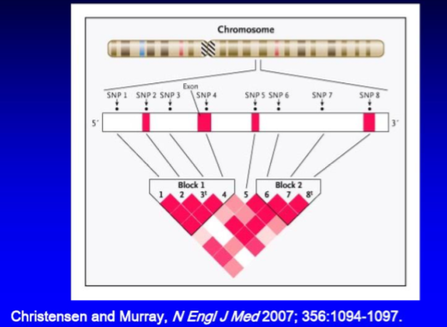
これを斜めにして、数字をとると以下のようになりますよね。

地理的な距離を、SNPs間の関連性に置き換えて考えると以下のようになります:
Spurious association(偽りの相関)について
偽りの相関とは、その名の通りでして、本体は関連がないのに、なんらかの理由で関係ありと判断されてしまった場合のことを言います。
- genotyping error
- bias
- type 1 error (False positive)
- population stratification
などが該当します。
Type I Errorについて
通常、統計学的な有意差というとα= 0.05を基準位しています。しかし、genome-wide association study(GWAS)のように、網羅的に検定を多数した場合も同じ水準でいると偽陽性(False positive)が増えてしまいます。このため、仮説検定の量だけ有意水準を下げる作業をします。
例えば100万の遺伝子を検定した場合に、Bonferroni adjustmentを行うとαは、
- α= 0.05/1,000,000 = 5 x 10-8
となります。
これまではBonferroni法がstandardとされてきましたが、保守的すぎるためfalse-positive discovery rate (FDR)やBayesian false discovery rateなどが使用されるケースもあります。
Hardy-Weinberg equilibriumからの逸脱(departure)
偽陽性の原因として、使用した遺伝子マーカーがHardy-Weinberg equilibriumからの逸脱している場合があります。
使用する遺伝子マーカーがHardy-Weinberg equilibriumに矛盾しないかをチェックする必要があります。
また、遺伝子マーカーそのものの問題でない場合もあり、
- DNAのcontamination
- 遺伝子検査の前の盲検化が不十分(information bias)
- genotyping error
なども原因になり得ます。
Population stratificationについて
あまり聞きなれない言葉かもしれませんが、”Population stratification(母集団の層別化)”が原因のこともあります。疫学で言えば交絡のことです。
例えば人種など母集団で遺伝子の構成・頻度は異なります。この場合は、実は検出された遺伝的な違いが疾患の原因であるわけではなく、単に母集団の違いが疾患に関連しているだけの可能性があります。
おわりに
今回はgenetic association studyの導入編的な内容を記載してきました。
Genome-wide association study(GWAS)は次回に解説できればと思います。
オススメの書籍はこちらになります。
最近、ミラーレス一眼レフを購入して、写真を楽しんでいます。
こちらのカメラを購入しましたが、綺麗に撮れて、持ち運びも楽、ミラーレスの中では非常に安いので、気に入っています。