今回は遺伝疫学的な考え方から、population stratificationとその対処法について解説していきます。
遺伝疫学を詳しく知りたい方は、以下の本をお勧めします↓
Population stratificationは、Race/ethnicityによる交絡
交絡や交絡因子についてわからない方は、以下の記事に詳しく書いているので、そちらを読んでみてください。
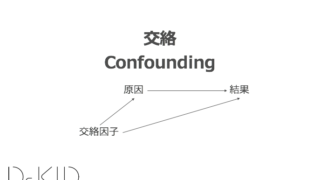
疫学研究でよく議論になる交絡(Confounding)と交絡因子(Confounder)ですが、遺伝疫学でも起こり得て、Population stratification confounding(集団の構造化による交絡)などと呼ばれています。
非常に簡単に説明すると、人種(race/ethnicity)による様々な生活様式・環境因子の違いから、交絡が生じてしまうことがあります。
DAGからPopulation stratificationを理解する
文章で解説するより、DAG(Directed Acyclic Graph)で記載した方が分かりやすいでしょう。
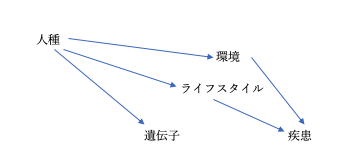
DAGが分からない方は、こちらの記事を読んでみてください。少なくとも疫学者の中では共通言語として使用していますので、一通り知っておくと良いでしょう。

本来は遺伝子と疾患の間に因果関係(Causation)はないのですが、人種による交絡で偽りの相関(Spurious association)が作り出されている状態を言います。
この状態を、Population stratificationなどと遺伝疫学では読んでいます。
Population stratificationを見分ける方法
Population stratificationを見つける際にQQ plotを使用することがあります。
QQ plotとは、quantile-quantile plotの略で、
- 計測されたP-valueをX軸に
- 予測されるP-valueをY軸に
プロットしたものです。P値はそのままの数字を使うのではなく、「–Log10」を使用します。これは、P-value = 1.0は帰無仮説と完全に矛盾しない状態をいい(perfect compatibility)、興味の対象ではありません。P値が小さいほど、「perfect compatibility」からより逸脱しているようにグラフで表示するために、–Log10」を使用しています。例えば、P = 1.0なら「–Log10(1.0) = 0」ですし、P = 0.001なら「–Log10(1.0) = 3」となります。
考え方としては、S-value(S値)に近いでしょう。
QQ plotの解釈の仕方ですが、大きく3つを知っておくと良いでしょう:
- 遺伝的な相関なし
- 遺伝的な相関があるかも
- バイアス混じり(Population stratificationなど)
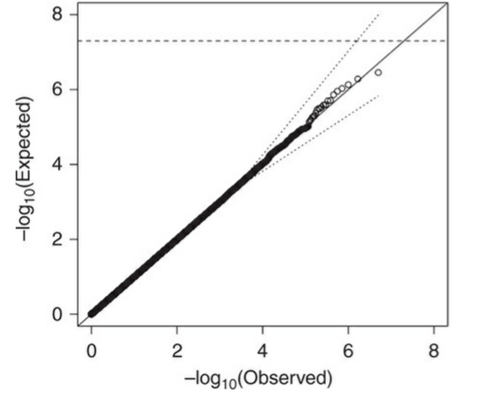
遺伝的な相関なし
遺伝的な相関がない場合、予測されるP値と計測されたP値が一致します。
この場合、QQ plotでは斜め45度の線に沿って表示されます。
遺伝的な相関があるかも
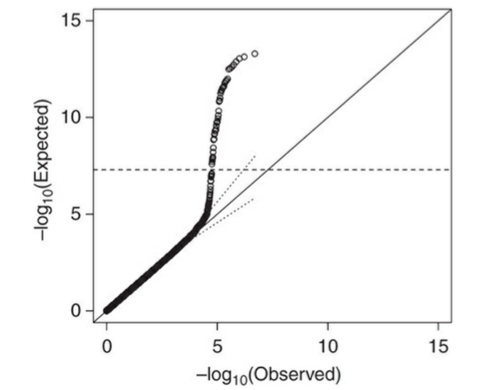
仮にpopulation stratificationなどのバイアスがない状態で以下のようなQQ plotを示した場合、遺伝的な相関が示唆される結果となります。
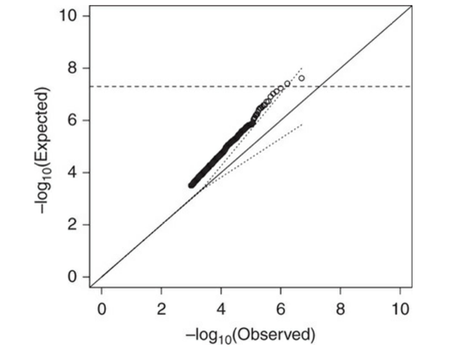
Population stratificationかもしれない
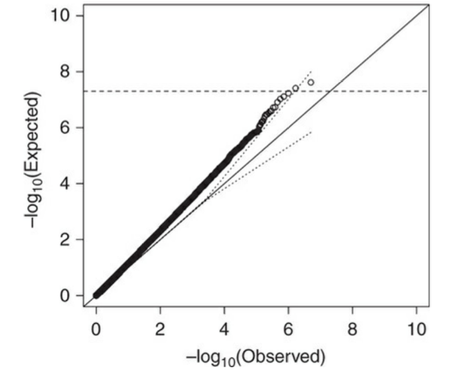
Population stratificationが示唆される場合として、以下のQQ plotが良い例です。
この場合、ほとんどのP値が、予測されるP値より低いことになります。
データの構造的な問題が示唆され、population stratificationを疑いたくなるような結果です。
QQ plotの問題点
QQ plotにもいくつか問題点があります。
その1つは、GWASのように大量のデータを使用する場合、コンピューターを使用したとしても非常に時間がかかってしまいます。
この対処法は2つあり、
- Hexagonal bin plottingを使用する
- 一定の値以下のP値を使用する
です。
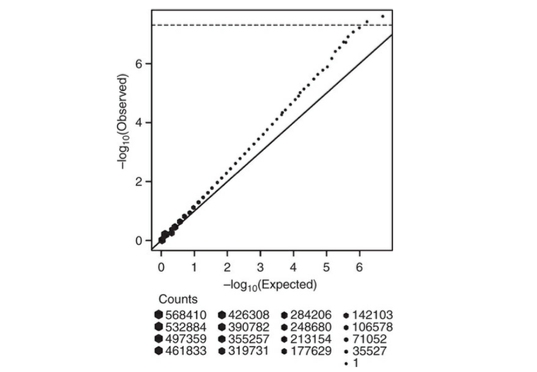
Hexagonal bin plottingについて
Hexagonal bin plotでは、1つ1つの値をプロットするのではなく、ある範囲にあるP値の値をまとめてしまい、それを六角形の大きさで図示してしまう方法です。
一定のP値以下のみをplotする
例えばP < 0.001のみをplotする方法もあります。
QQ plotにおいて左下の情報はそれほど重要ではないため、この場所を節約する試みとも言えます。
Population stratificationを対処する方法
Population stratificationを対処する方法はいくつかあり、
- Genomic control
- Ethnicity adjustment
- Principal component adjustment
- Estimated ancestry adjustment
などがあります。今回は、Genomic controlとEthnicity adjustmentについて解説します。
Genomic controlについて
Genomic controlですが、その名の通り「コントロールとなる遺伝子(SNP)」を使用して行う解析です。
つまり、「おそらく疾患には関与しないだろうと思われる遺伝子やSNP」を使用して、実際に計測された分布と比較します。
このgenomic-controlλは、
λ= [計測されたtest statisticsの中央値]/ [Chi-square分布での中央値]
の比を計算します。
この比が1.00〜1.05より大きければpopulation stratificationが示唆され、この値より小さければ可能性は低いと判断します。
Genomic controlの問題点
Genetic controlの問題点として、特にGWASなど網羅的に解析する場合に、どの遺伝子をコントロールとすべきか不明なことが多いためです。一方で、とある遺伝子と疾患の関連性を検討する時、ほとんどのSNPは関連していません。
また、population stratificationの数が多い場合、対処し切れていない場合もあります。
さらには、genetic controlの結果は保守的であるため、偽陰性の可能性が高いとも言われています
Ethnicity adjustmentについて
Ethnicity adjustmentとは、Ethnicity(人種など)のデータを用いて、統計学的に対処する方法です。
例えばアウトカムがY、人種がX、他の交絡因子がZ、Gが遺伝子の情報であれば、
f(Y) = β0 +β1X + β2Z + β3G
と統計学的なモデルを組むことができます。
そして、β3が人種や交絡因子を対処した後に認められた遺伝子が疾患に与える影響と言えます。
Ethnicity Adjustmentの問題点
問題点としては、race/ethnicityのデータがそもそも揃っていない場合、この解析を行うことができないことです。
さらには、人種や民族によっては、admixture(混合)を起こしていることがあります。
この考え方と対処法については次回に説明します。
遺伝疫学を詳しく知りたい方は、以下の本をお勧めします↓